랜덤 포레스트를 위한 2가지 R 함수 party::cforest vs. randomForest::randomForest 비교하기
(1) {party} cforest를 사용하여 랜덤 포레스트 구현하기
(2) {randomForest} randomForest를 사용하여 랜덤 포레스트 구현하기
(3) 두 함수의 성능 비교하기
랜덤 포레스트를 구현하기 위한 R 패키지와 함수는 크게 2가지가 있다.
Ⅰ party 패키지의 cforest 함수로
Ⅱ. randomForest 패키지의 randomForest 함수 (caret 패키지를 이용하는 경우에도 이 함수를 사용하는 것과 같다.)
이번 포스팅에서는 두 함수를 사용하는 방법과 두 함수를 이용해 Variable Importance를 구하는 방법, 그리고 성능을 비교해보고자 한다.
사용할 데이터는 R 기본 제공 데이터인 iris이며, iris의 Sepal.Length, Sepal.Width, Petal.Length, Petal.Width를 이용해 Species 3가지 중 어떤 Species인지 구분하도록 랜덤 포레스트 알고리즘을 구현하는 코드를 작성해 보겠다.
(1) {party} cforest를 사용하여 랜덤 포레스트 구현하기
1. 먼저 R 기본 제공 데이터인 iris 데이터를 불러온다.
data(iris)2. 패키지 설치하고 라이브러리 불러온다.
cforest 함수를 사용할 수 있도록 party 패키지를 설치하고 라이브러리를 불러온다.
install.packages("party")
library(party)3. cforest 함수를 이용해 랜덤 포레스트 모델을 생성한다.
랜덤 포레스트는 이름 그대로 "Random"성이 있기 때문에 set.seed 함수를 이용해 재현할 수 있도록 구현한다.
옵션으로는 mtry = 2 (하나의 트리에 2개의 변수를 선택해 트리를 구성)와 ntree = 100 (이런 트리를 100개 만들도록) 옵션을 설정했다.
set.seed(1)
cf <- cforest(formula = Species ~ .,
data = iris,
controls = cforest_unbiased(mtry = 2, ntree = 100))
cf4. Variable Importance를 추출한다.
랜덤 포레스트를 사용하는 경우는 주로 Variable Importance를 추출하기 위한 경우가 많은 것 같다. 랜덤 포레스트 함수들은 반응변수(여기서는 Species)를 분류할 때 가장 영향을 끼치는 변수가 무엇인지 판단할 수 있는 수치를 제공한다.
cforest 함수에서 Variable Importance를 추출하기 위해서는 varimp 함수를 이용한다.
cf_varimp <- varimp(cf)
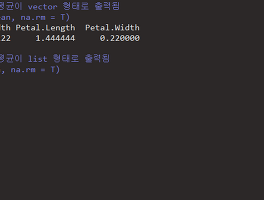
cf_varimp결과를 해석하자면 iris 데이터에서 Species를 분류할 때 가장 중요한 역할을 하는 변수는 Petal.Width > Petal. Length > Sepal.Length > Sepal.Width 순이라는 의미이다.
이번에는 randomForest 패키지의 randomForest 함수를 사용해서 위와 같은 코드를 작성해보자.
(2). {randomForest} randomForest를 사용하여 랜덤 포레스트 구현하기
1. 먼저 R 기본 제공 데이터인 iris 데이터를 불러온다.
data(iris)2. 패키지 설치하고 라이브러리 불러온다.
randomForest 함수를 사용할 수 있도록 randomForest 패키지를 설치하고 라이브러리를 불러온다. (대소문자에 주의한다.)
install.packages("randomForest")
library(randomForest)3. randomForest 함수를 이용해 랜덤 포레스트 모델을 생성한다.
랜덤 포레스트는 이름 그대로 "Random"성이 있기 때문에 set.seed 함수를 이용해 재현할 수 있도록 구현한다.
위에서 작성한 cforest와 동일하게 옵션으로 mtry = 2 (하나의 트리에 2개의 변수를 선택해 트리를 구성)와 ntree = 100 (이런 트리를 100개 만들도록) 옵션을 설정했다.
set.seed(1)
rf <- randomForest(formula = Species ~ .,
data = iris,
mtry = 2,
ntree = 100,
corr.bias = T)
rfrandomForest 함수의 경우 결과 출력시 Confusion Matrix로 Classification Error Rate까지 출력해준다.
4. Variable Importance를 추출한다.
randomForest 함수에서 Variable Importance를 추출하기 위해서는 importance 함수를 이용한다.
rf_varimp <- importance(rf, type = 2)
rf_varimp결과를 해석하자면 iris 데이터에서 Species를 분류할 때 가장 중요한 역할을 하는 변수는 Petal.Width > Petal. Length > Sepal.Length > Sepal.Width 순이라는 의미이다. 이 결과는 앞서 cforest를 이용한 Variable Importance 추출 결과와 비교할 때 수치 자체는 다르지만 결과는 변수 중요도 순서는 동일하다.
하지만 변수의 수가 많은 경우, set.seed를 설정하지 않으면 다양한 랜덤 포레스트 결과가 생성되기 때문에 변수 중요도 순서와 수치는 계속 변경될 수 있다는 점을 꼭 명심하자.
이번에는 두 Random Forest 함수를 돌려 Variable Importance를 추출하는데 까지 걸리는 시간을 비교해보려고 한다.
(3) 두 함수의 성능 비교하기
여기서 말하는 성능이란, 처리 결과와 처리 시간을 말한다. 처리 결과와 처리 시간을 비교하는 실험을 설계해보자.
1) 데이터는 iris 데이터를 10배 뻥튀기한 데이터를 사용한다.
2) Species 각 종별 100건, 250건, 500건씩 추출하여 데이터의 row 수는 300, 750, 1500 3가지 단계로 비교한다.
3) set.seed를 설정하지 않고 랜덤으로 데이터 사이즈당 5번씩 반복한다.
1. 먼저 위에 설계한대로 결과 저장을 위한 데이터프레임을 생성한다.
tm_result는 처리시간 테스트 결과를 저장하는 데이터프레임이고, rk_result는 Variable Importance 처리결과의 수치를 저장하는 데이터프레임이다.
tm_result <- data.frame(size = rep(c(300, 750, 1500), each = 5),
k = rep(c(1:5), 3),
cf_time = NA,
cf_unit = NA,
rf_time = NA,
rf_unit = NA)
rk_result <- data.frame(size = rep(c(300, 750, 1500), each = 10),
k = rep(c(1:5), each = 2, 3),
func = rep(c("cf", "rf"), 15),
Sepal.Length = NA,
Sepal.Width = NA,
Petal.Length = NA,
Petal.Width = NA)2. 처리시간 테스트를 위한 반복문을 작성한다.
처리시간 테스트에는 부하를 주기 위해 cforest, randomForest 각각 트리를 3000개씩 생성하도록 ntree 옵션을 변경했다.
# 처리시간 테스트
for(n in c(10, 25, 50)){
data <- iris[c(1:(1+n-1), 51:(51+n-1), 101:(101+n-1)), ]
dat <- rbind(data, data, data, data, data, data, data, data, data, data)
# 반복문 1~5
for(k in 1:5){
dtm <- Sys.time()
cf <- cforest(formula = Species ~ .,
data = dat,
controls = cforest_unbiased(mtry = 2, ntree = 3000))
cf_varimp <- varimp(cf)
time <- Sys.time() - dtm
tm_result[tm_result$size == nrow(dat) & tm_result$k == k, "cf_time"] <- time
tm_result[tm_result$size == nrow(dat) & tm_result$k == k, "cf_unit"] <- attributes(time)$units
dtm <- Sys.time()
rf <- randomForest(formula = Species ~ .,
data = dat,
mtry = 2,
ntree = 3000,
corr.bias = T)
rf_varimp <- importance(rf, type = 2)
time <- Sys.time() - dtm
tm_result[tm_result$size == nrow(dat) & tm_result$k == k, "rf_time"] <- time
tm_result[tm_result$size == nrow(dat) & tm_result$k == k, "rf_unit"] <- attributes(time)$units
}
}
3. 처리시간 테스트 결과를 해석한다.
먼저 Sys.time 함수의 숫자는 단위에 따라 크기가 다르기 때문에 단위를 sec로 맞춰준다.
tm_result$cf_time <- ifelse(tm_result$cf_units == "mins", tm_result$cf_time * 60, tm_result$cf_time)
tm_result$rf_time <- ifelse(tm_result$rf_units == "mins", tm_result$rf_time * 60, tm_result$rf_time)library(plyr)
tm_summary <- ddply(tm_result, "size", summarise, avg_cf = mean(cf_time), avg_rf = mean(rf_time))
평균 처리시간을 선 그래프로 시각화한다.
library(scales)
plot(formula = avg_cf ~ size,
data = tm_summary,
type = "o",
col = alpha("black", 0.5),
xlim = c(200, 1500),
ylim = c(0, max(tm_summary$avg_cf)),
xlab = "n rows",
ylab = "sec",
main = "cforest vs. randomForest")
lines(formula = avg_rf ~ size,
data = tm_summary,
type = "o",
col = alpha("red", 0.5))
legend("topleft",
c("avg_cf", "avg_rf"),
pch = 1,
col = alpha(c("black", "red"), 0.5),
cex = 0.9,
bty = "n")위 그래프를 보면 데이터의 건수가 늘어날 수록 randomForest 대비 cforest의 처리속도가 기하급수적으로 늘어나는 것을 확인할 수 있다. 따라서 처리 속도 측면에서 보았을 때는 randomForest 패키지의 randomForest 함수가 더 유리하는 것을 알 수 있다.
4. 처리결과 테스트를 위한 반복문을 작성한다.
4개의 변수에 대한 중요도 점수를 결과 데이터프레임에 입력하도록 반복문을 작성한다.
# 처리결과 테스트
for(n in c(10, 25, 50)){
data <- iris[c(1:(1+n-1), 51:(51+n-1), 101:(101+n-1)), ]
dat <- rbind(data, data, data, data, data, data, data, data, data, data)
# 반복문 1~5
for(k in 1:5){
cf <- cforest(formula = Species ~ .,
data = dat,
controls = cforest_unbiased(mtry = 2, ntree = 100))
cf_varimp <- varimp(cf)
rk_result[rk_result$size == nrow(dat) & rk_result$k == k & rk_result$func == "cf", "Sepal.Length"] <- as.numeric(cf_varimp["Sepal.Length"])
rk_result[rk_result$size == nrow(dat) & rk_result$k == k & rk_result$func == "cf", "Sepal.Width"] <- as.numeric(cf_varimp["Sepal.Width"])
rk_result[rk_result$size == nrow(dat) & rk_result$k == k & rk_result$func == "cf", "Petal.Length"] <- as.numeric(cf_varimp["Petal.Length"])
rk_result[rk_result$size == nrow(dat) & rk_result$k == k & rk_result$func == "cf", "Petal.Width"] <- as.numeric(cf_varimp["Petal.Width"])
rf <- randomForest(formula = Species ~ .,
data = dat,
mtry = 2,
ntree = 100,
corr.bias = T)
rf_varimp <- importance(rf, type = 2)
rk_result[rk_result$size == nrow(dat) & rk_result$k == k & rk_result$func == "rf", "Sepal.Length"] <- rf_varimp["Sepal.Length", ]
rk_result[rk_result$size == nrow(dat) & rk_result$k == k & rk_result$func == "rf", "Sepal.Width"] <- rf_varimp["Sepal.Width", ]
rk_result[rk_result$size == nrow(dat) & rk_result$k == k & rk_result$func == "rf", "Petal.Length"] <- rf_varimp["Petal.Length", ]
rk_result[rk_result$size == nrow(dat) & rk_result$k == k & rk_result$func == "rf", "Petal.Width"] <- rf_varimp["Petal.Width", ]
}
}결과 테이블 중 데이터 사이즈가 300일 때를 살펴보자.
cforest의 경우 1회차, 4회차에는 Petal.Length가, 2회차, 3회차, 5회차에는 Petal.Width가 가장 높은 점수를 받은 것을 알 수 있다.
randomForest의 경우 1회차부터 5회차까지 모두 Petal.Width가 높은 점수를 받은 것을 알 수 있다.
이 결과만 봤을 때는 처리결과 측면에서도 randomForest가 더 일관성이 있는 것처럼 보여서 랜덤 포레스트를 R로 구현할 때에는 party 패키지의 cforest보다는 randomForest 패키지의 randomForest 함수를 사용하는 것이 더 좋은 것 같다.
'회사생활 > R' 카테고리의 다른 글
| R apply 계열 함수 총 정리 1 ( apply / lapply / sapply / vapply ) (2) | 2017.06.25 |
|---|---|
| 한눈에 정리하는 ggplot2를 이용한 R 시각화 기초 2 (0) | 2017.04.26 |
| 로컬에서 데이터 불러오기 / 데이터 저장하기 (0) | 2017.03.26 |
| [R 예제 코드] Logistic Regression / 로지스틱 회귀분석 (19) | 2017.03.22 |
| 한눈에 정리하는 ggplot2를 이용한 R 시각화 기초 1 (2) | 2017.03.17 |